L'ADN fossile
On appelle ADN fossile, l’ADN retrouvé dans les restes fossiles. L’intérêt de l’ADN fossile est de récupérer des traces génétiques d’espèces disparues dans leurs fossiles. Elle est notamment la principale source de la recherche d’ADN pour cloner les espèces éteintes
La récupération de l'ADN fossile pose cependant de nombreux problèmes. L’ADN ancien est certes une molécule stable, mais elle n'est pas éternelle. Sa dégradation commence même aussitôt la mort d'un individu survenue ; les membranes de cellules cèdent, les tissus se désorganisent et baignent dans leurs propres liquides. Des dégradations chimiques se produisent :
La dégradation chimique : elle se traduit soit par une hydrolyse (provoquant une rupture de la chaîne d’ADN) ; soit par une oxydation (ayant pour conséquences l’endommagement de l’ADN et l’impossibilité des nucléotides complémentaires de se lier entre eux).
La dégradation enzymatique : Les enzymes de ce dernier vont digérer l’ADN et les molécules restantes, c’est ce que l’on appelle l’autolyse. Cela entraînera la décomposition des tissus et donc la putréfaction du cadavre.
Résultat : les molécules d'ADN se fragmentent et certaines des lettres constitutives de l'information génétique sont peu à peu effacées. Les chimistes estiment que dans ces conditions, une dizaine de milliers d'années peut suffire à venir à bout de la dernière molécule d'ADN. Ajoutez à cela les effets des organismes décomposeurs. Ainsi il n'y a presque chance de retrouver de l'ADN à des époques aussi lointaines que celles des dinosaures. Nous pourrions remonter qu’à quelques dizaines de milliers d'années tout au plus. À moins que des conditions environnementales n'aient considérablement ralenti les réactions de dégradations car alors peut-on atteindre à l'extrême limite quelques centaines de milliers d'années. La bonne conservation de l'ADN dans les restes fossiles dépend des facteurs physico-chimiques environnementaux (pH, température, humidité, pression) qui interagissent entre eux de manière complexe. Le facteur temps ne semble pas, quant à lui, être un paramètre fondamental. Les dégradations chimiques et enzymatiques sont ralenties lorsque les températures sont faibles – l'idéal étant la congélation – ou dans des milieux anaérobies. Les milieux froids, les déserts chauds et secs, les tourbières et les fosses à goudron préservent donc mieux l'ADN ancien. De telles ressources sont donc à rechercher du côté des spécimens congelés dans les glaces de Sibérie ou du Groenland ou les pergélisols péri-arctiques.
Techniquement, récupérer de l'ADN à partir d'un reste fossile est une opération extrêmement délicate. Tout d'abord, ce qui reste de l'ADN de départ est mal-en-point. Notre ADN est infiniment en meilleur état. L’ADN ancien est de plus sujet à de nombreuses contaminations par de l’ADN moderne. Celle-ci peut avoir des sources variées et intervenir à différents niveaux; elle peut être naturelle (propre au spécimen comme en cas de présence de parasites ou de micro-organismes au moment de la mort), ou ultérieure à la mort (lors de la découverte du spécimen ou en laboratoire pendant l'extraction ou l'amplification). De nombreuses personnes manipulent les restes entre la mort de l'organisme étudié et l'analyse en laboratoire, et les contaminations sont donc difficiles à contrôler. Il est notamment conseillé de porter des gants stériles pour tous les prélèvements et de disposer, dans le laboratoire, de pièces isolées, réservées à l'étude de l'ADN ancien, et une atmosphère maintenue en surpression, en séparant les différents postes de travail et en imposant un sens de manipulation strict, afin d'éviter toute entrée indésirable d'ADN depuis l'extérieur. Les surfaces sont nettoyées quotidiennement à l'eau de javel et en permanence exposées aux rayons ultraviolets. Toutes ces précautions visent à minimiser la part des contaminations par l'ADN actuel.
Voyons maintenant le protocole opératoire. Il faut d’abord broyer un échantillon d'environ cinq cent milligrammes d'un fossile. Ensuite, la poudre est mise à incuber plusieurs heures dans un tampon facilitant la dégradation des matériaux indésirables (protéines par exemple), et s'il s'agit de la poudre d'os ou de dents, d'un agent dissociant la matrice calcifiée. Résultat : l'ADN, s'il est encore préservé, est libéré dans le tampon. La suite des opérations ne vise donc plus qu'à le précipiter et concentrer dans un volume facilitant sa manipulation. Cependant, à ce stade, ne subsistent que de rares traces d'ADN en mauvais état, soit autant de mauvais substrat relativement réfractaires aux techniques classiques de séquençage. Il faut donc généralement viser une région précise du génome et la photocopier des milliards de fois avant d'espérer déterminer la séquence souhaitée.
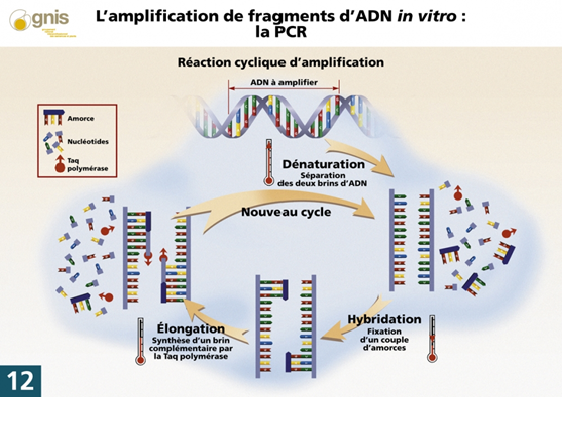 C'est précisément l'objet de la PCR (Polymerase Chain Reaction, ou Réaction de Polymérisation en Chaîne), une technique de biologie moléculaire mise au point au milieu des années 1980, et désormais courante dans les laboratoires. Son principe repose sur l'utilisation d'une enzyme qui synthétise une chaîne d'ADN à partir de précurseurs des nucléotides. L'opération est répétée un certain nombre de fois et, le résultat de cette opération étant exponentiel, on obtient ainsi un grand nombre de molécules d'ADN identiques à la molécule initiale. La caractéristique majeure de la P.C.R. est son extrême sensibilité (en théorie une molécule initiale suffit). Cependant, si la technique présente l'avantage de ramener en quantités suffisantes des substrats qui ne subsistaient plus qu'à l'état de traces, la technique peut, si l'on n'y prend garde, tout aussi bien multiplier les contaminants. L'étape est donc particulièrement risquée et délicate et nécessite de nombreux contrôles de qualité. Les molécules d'ADN fossile étant particulièrement abîmées, les photocopier engendre de très nombreuses coquilles. Il faudra donc encore dépouiller les exemplaires photocopiés de ces coquilles avant de recomposer la séquence finale. Et gage ultime d'authenticité, la séquence doit être reproduite plusieurs fois, et de préférence, dans un laboratoire indépendant.
C'est précisément l'objet de la PCR (Polymerase Chain Reaction, ou Réaction de Polymérisation en Chaîne), une technique de biologie moléculaire mise au point au milieu des années 1980, et désormais courante dans les laboratoires. Son principe repose sur l'utilisation d'une enzyme qui synthétise une chaîne d'ADN à partir de précurseurs des nucléotides. L'opération est répétée un certain nombre de fois et, le résultat de cette opération étant exponentiel, on obtient ainsi un grand nombre de molécules d'ADN identiques à la molécule initiale. La caractéristique majeure de la P.C.R. est son extrême sensibilité (en théorie une molécule initiale suffit). Cependant, si la technique présente l'avantage de ramener en quantités suffisantes des substrats qui ne subsistaient plus qu'à l'état de traces, la technique peut, si l'on n'y prend garde, tout aussi bien multiplier les contaminants. L'étape est donc particulièrement risquée et délicate et nécessite de nombreux contrôles de qualité. Les molécules d'ADN fossile étant particulièrement abîmées, les photocopier engendre de très nombreuses coquilles. Il faudra donc encore dépouiller les exemplaires photocopiés de ces coquilles avant de recomposer la séquence finale. Et gage ultime d'authenticité, la séquence doit être reproduite plusieurs fois, et de préférence, dans un laboratoire indépendant.

Long et périlleux est donc le chemin qui mène à une séquence d'ADN fossile. Et pourtant, combien cette dernière est courte ; en effet, dans les fossiles, l'ADN n'est pas conservé sous la forme de longs chromosomes intègres mais sous la forme de très courts fragments. Aussi ne peut-on généralement espérer obtenir suite à une photocopie qu'un fragment long de cent cinquante à deux cents nucléotides. Inutile de préciser que dans ces conditions, plusieurs fragments seront nécessaires avant d'obtenir une quantité d'information suffisante pour répondre aux questions posées. Autre point très important : le plus souvent, les gènes ciblés ne correspondent pas à des gènes portés par les chromosomes. Ils sont hébergés par un autre type d'ADN qui dans nos cellules, n'est pas contenu dans le noyau mais dans d'autres compartiments : les mitochondries (il s'agit donc de l'ADN mitochondrial). C'est au sein des mitochondries que s’opèrent les réactions de respiration dans nos cellules. Certes, cet ADN-là est-il généralement court - environ seize mille nucléotides chez l'Homme – mais il présente plusieurs avantages : d'une part, son évolution est rapide si bien qu'il sera possible de suivre des événements du passé avec une bonne résolution et d'autre part, chaque cellule présente au bas mot un millier de mitochondries. Autant dire, un millier de molécules d'ADN mitochondrial. Or, chaque chromosome dans notre noyau n'est présent qu'en deux exemplaires. Ainsi, en ciblant spécifiquement l'ADN mitochondrial, les paléogénéticiens multiplient les chances de réussir à mettre la main sur un bout d'information encore conservée.

Ainsi, il est peu probable de trouver une quantité convenable d’ADN viable qui nous permettrait d’envisager le clonage. Autre fait : Des chercheurs ont établi que la demi-vie de l’ADN était de 521 ans, c’est à dire qu’après ces 521 années, la moitié de la molécule est dégradée. Pour aller plus loin, ces mêmes chercheurs ont alors estimé qu’après environ 6.8 millions d’années, même en conditions de conservation optimales, toute la molécule serait dégradée et que même après 1,5 million d’années, le peu d’ADN présent serait déjà quasiment inexploitable.
Il s’agit là d’un obstacle apparemment infranchissable pour certaines espèces éteintes comme les dinosaures. En effet, ces derniers se sont, pour les moins vieux, éteints en masse il y a 65 millions d’années, soit il y a 10 fois plus longtemps que la durée de vie maximale proposée pour la molécule d’ADN. En d’autres mots, il semble du domaine de l’impossible de récupérer un bout d’ADN viable de dinosaure. Récupérer de l’ADN d’espèces éteintes est une tâche très difficile. Afin d’obtenir de l’ADN